品牌
其他厂商性质
所在地

RAD SOURCE R1800Q 细胞辐照仪
面议
Naolive SA 3D Cell Explorer无标记活细胞3D显微镜
面议
Nicoyalife OpenSPR分子互作仪
面议
Protein Marker高通量蛋白纯化系统
面议
德国美诺实验室清洗消毒机(全自动洗瓶机)耗材配件
面议
德国美诺Miele PG8583系列实验室清洗消毒机(全自动洗瓶机)
面议
Rad Source RS34000 X射线*
面议
ThermoFisher CellInsight CX7高内涵筛选(HCS)平台
面议
ThermoFisher CellInsight CX5高内涵筛选(HCS)平台
面议
MD ImageXpress Pico 个人型高内涵成像分析系统
面议
SCANCO vivaCT 80 小动物临床前扫描系统
面议
AES CEInfinite全柱成像等电聚焦毛细管电泳系统
面议
一个系统,一套工作流程,强大的测序应用 10xGenomics公司推出了的Chromium™系统。利用上千万*的DNA序列标记的GEMs,Chromium™系统可揭示重要的基因组信息。
1、全功能仪器10xChromiumController具有可伸缩的托盘和触屏界面,占用空间极小,不超过1平方英尺,却能够高效的将10万到100万次的移液步骤自动化,采用*的微流控系统,高通量的对样品进行分离并带上序列标签,终达到高通量检测的目的。10xChromiumController可以进行基因组的分析,同时可以进行单细胞水平的功能分析。
2、单细胞仪器10xChromiumSingleCellController具有可伸缩的托盘和触屏界面,占用空间极小,不超过1平方英尺,却能够高效的将10万到100万次的移液步骤自动化,采用*的微流控系统,高通量的对样品进行分离并带上序列标签,达到高通量检测的目的。10xChromiumSingleCellController只可以进行单细胞水平的功能分析。

提升组装参数:结合该技术的组装效果较单独使用Illumina数据可提升十几倍;
测序平台通用性:应用该技术构建的文库可与Illumina测序系统完美匹配;
分子片段长:分析可将短Reads组装成长达50-100Kb的linked-reads;
单倍体分析:可实现染色体级别Phasing,获得精确的单体型信息。

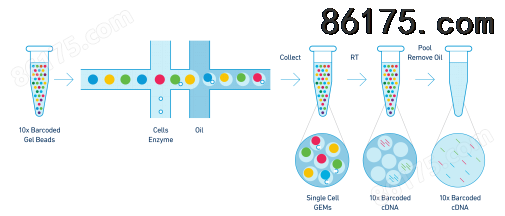
1. 将样本分配到100,000s到1000,000s微反应体系,每个微反应体系含有一种特定的DNA序列标记。
2.含有barcode信息的凝胶珠子首先与样品和酶的混合物混合,然后与位于微流体“双十字”连接中的油表面活性剂溶液结合。
3.GEMs (包含样品、酶、凝胶珠子的混合物油滴)流到储液器中并进行收集。凝胶珠子溶解释放barcode序列,开始对样本进行标记。将每个液滴中含有barcode信息的产物混合。然后构建标准测序文库。
1.基因组组装:利用GemCode平台对长片段序列进行扩增引入barcode序列以及测序接头引物,然后将序列打断成适合测序大小的片段进行测序,通过barcode序列信息将多个Reads进行组装,获得跨度在30-100Kb的linked-reads信息,从而获得大片段的遗传信息。
优势:
通过该技术构建的文库可与Illumina测序系统完美匹配,将短Reads组装成长达100Kb的片段;
结合该技术后组装效果较单独使用Illumina数据可提升十几倍;
精度高、成本低,操作难度小。
2.单细胞转录组测序:基于10x GenomicsChromium™系统利用油包水的微反应体系,通过序列标签区别群体中的不同细胞,获得单细胞水平的数字化基因表达谱,可实现数千甚至数万个单细胞群体分析,解决常规scRNA-seq方法在通量或扩展性方面存在的不足,为单细胞研究开拓新的思路。
优势:
超高通量:单个样本细胞检测数可达1000-10000;
项目周期短:一天内即完成从细胞悬液到cDNA文库构建所有的测序准备工作;
细胞捕获效率高:单个细胞捕获效率高达65%,可准确鉴别稀有细胞类型;
真正的单细胞测序:通过油滴-barcode-单细胞的对应关系,实现真正意义上的单细胞测序;
测序平台兼容度广:与Illumina各种型号的平台高度兼容。


gDNA
片段要求:主带﹥50kb 50kb~100kb插入片段 Illumina PE150
测 序深度≥100X
应用10x Genomics辅助基因组组装效果评估
目前对人类基因组de novo测序和组装的成功案例是将Illumina短片段测序、PacBio单分子实时测序技术、BioNano光学图谱技术相结合,但这种方法中PacBio的测序成本相对较高。
本研究提供了一种基因组de novo测序和组装的新方法。这种方法使用10x Genomics的linked-reads来获得中长片段的数据,然后使用Illumina测序,结合BioNano构建基因组图谱,对contigs进行anchoring和scaffolding,终组装的基因组Scaffold N50达33.5Mb。并通过对人类HapMap样品(NA12878)进行基因组de novo组装验证了这种方法的可行性。
使用10x Genomics+Illumina+BioNano的方法进行基因组组装的结果表明,scaffold N50长度和组装的基因组长度均显著提高,而scaffold的数目显著减少。
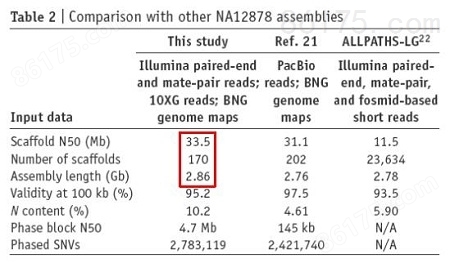
Yulia Mostovoy, Michal Levy-Sakin, et al., A hybrid approach for de novo human genome sequence assembly and phasing. Nature Methods. 2016 May 9. doi: 10.1038/NMETH.3865.